Desde la rueda hasta la turbina la humanidad se ha inventado un buen número de máquinas para realizar trabajos mecánicos muy diversos, por lo que parece natural que la misma función, como impulsar un coche, pueda hacerse con motores de diseños muy diferentes. También en las distintas células se puede hacer una misma función utilizando maquinarias de diferentes diseños. Así como la mayoría de los coches utilizan un motor de gasolina, la mayoría de los microbios y muchos orgánulos como los cloroplastos, para llevar a cabo la división en dos, utilizan un elemento común, la proteína FtsZ.

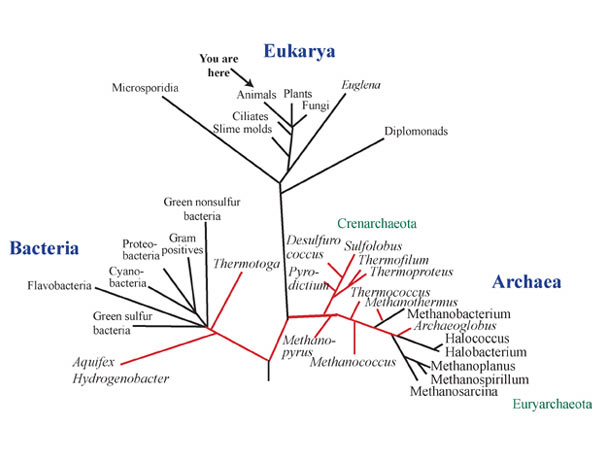
Distribución filogénetica simplificada de la proteína FtsZ.
Esta proteína es imprescindible para la división celular de un gran número de bacterias, arqueas y orgánulos.
Sin embargo hay vehículos que en vez de un motor de gasolina usan un motor eléctrico o incluso una caldera a vapor como las locomotoras. De hecho hay microbios que prescinden por completo de FtsZ y utilizan otro sistema para realizar su división. Ya se sabía que algunos parásitos intracelulares, como Chlamydia, carecen de dicha proteína. ahora se les une un importante grupo de arqueas, las Crenarchaeotas a las que pertenece Sulfolobus acidocaldarius, la especie en la que se ha hecho el descubrimiento.
Un hogar muy especial.
El jardín de las delicias de Sulfolobus acidocaldarius para nosotros sería mas bien un infierno, casi hirviendo a 80ºC y bastante ácido a pH 2. Su hogar, en las solfataras, se asemeja asombrosamente a las calderas de Pedro Botero ilustradas en el Hortus deliciarum, un manuscrito medieval compilado por Herrad def Landsberg entre 1167 y 1185.
¿Qué es y qué hace FtsZ?
FtsZ es una proteína con estructura y propiedades muy parecidas a la Tubulina, la proteína que en las células eucariotas como las humanas, forma los microtúbulos del citoesqueleto. Estos microtúbulos son los encargados de procesos tan importantes como el tráfico de vesículas por el citoplasma y del reparto de los cromososmas durante la mitosis. En la división de bacterias y de orgánulos de origen bacteriano, FtsZ se coloca en el centro de la célula y dirige el ensamblaje de todos los componentes que integran un anillo que ejecuta la constricción de la membrana celular. FtsZ es una proteína esencial, es decir que si una bacteria no la puede producir queda condenada a crecer sin poder dividirse, lo mismo que le ocurre a un cloroplasto privado de ella.

Estructura tridimensional de FtsZ y Tubulina.
Pese a que funcionan en procesos diferentes, las dos proteínas tienen una gran similitud estructural, que reside no tanto en los aminoácidos que componen sus secuencias, sino en el tipo de estructuras en hélice y lámina que adoptan.
¿Cómo lo hacen algunas arqueas?
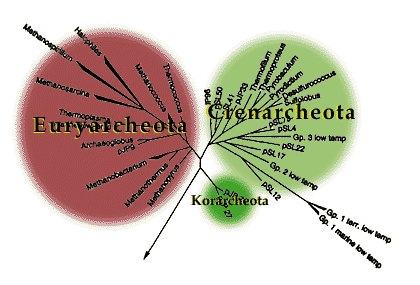
Ahora mismo se reconoce que los microorganismos pertenecientes al Dominio Archaea están divididos en dos tipos distintos o phyla: Crenarchaeota y Euryarchaeota, aunque es probable que dentro de poco haya más tipos. Los euryarqueotas incluyen a los metanógenos y a las arqueas de ambientes hipersalinos mientras que los crenarqueotas son microorganismos termófilos e hipertermófilos. Pues bien, sólo los euryarqueotas parecían utilizar un mecanismo de división celular basado en el anillo contráctil de la proteína FtsZ. Es el caso de la arquea halófila Haloferax volcanii.
La división sin Z.
Por eso resultaba chocante no encontrar FtsZ, o algo muy similar, en las crenarqueas. Un grupo de investigadores de la Universidad de Uppsala, ha resuelto ahora este enigma: las crenarqueas sin FtsZ, han montado su maquinaria de división basándose en proteínas por completo diferentes a las de las bacterias y a las de sus primas las euryarqueas. Estas proteínas muestran homología con un tipo de proteínas eucariotas. La sorpresa es que no se parecen para nada a la Tubulina, y tampoco a la actina o la miosina, proteínas que participan en la división de eucariotas. Se parecen a las proteínas involucradas en la formación de vesículas endosomales a partir del retículo endoplasmático, o en la liberación de virus como el VIH.
Rolf Bernander y sus colaboradores han identificado tres proteínas de Sulfolobus acidocaldarius, denominadas CdvA, CdvB y CdvC, que se producen en el momento correcto en el que se predice comienza la división. Además, y como ocurre con varias de las proteínas que las bacterias utilizan para dividirse, los genes que las codifican están pegados en el mismo lugar del genoma.
Se les ha llamado operón cdv, un acrónimo de "celular-división". Dentro del trío, las proteínas CdvB y CdvC, son las que tienen homología con las proteínas eucariotas del complejo ESCRT-III. Dicho complejo forma vesículas en el lumen de las vesículas endosomales por un proceso conocido como “círculos concéntricos”. Si nos paramos un momento a pensar, crear una vesícula es algo parecido a una división celular por gemación. El tercer gen, cdvA, tiene la información para formar una proteína que se parece a otras proteínas del citoesqueleto eucariótico distintas a la Tubulina.

Parientes lejanos
En el esquema de la izquierda se representan las fases finales de la división de Sulfolobus acidocaldarius. Tras la replicación y segregación de los cromosomas se forma una constricción en el centro de la célula. Se ha observado que las proteínas Cdv se disponen en esa zona central y una hipótesis plausible es que intervienen activamente en dicha constricción. A la derecha se muestra el proceso de formación de vesículas endosomales en las rutas de procesamiento de los receptores de membrana de las células eucariotas. Dichos receptores son internalizados en la célula mediante una invaginación de la membrana plasmática formando una vesícula, la cual une a otras semejantes formando una vesícula más grande llamada endosoma. El endosoma sufre a su vez nuevas invaginaciones, liberándose pequeñas vesículas en su interior. Es en esa segunda invaginación donde están involucradas las proteínas pertenecientes al complejo ESCRT-III (etapas 1, 2 y 3). El resultado es que se forma una gran vesícula llena a su vez de vesiculitas conocida como cuerpo multivesicular, que acaba fusionándose con un lisosoma para la degradación de su contenido.
La expresión de los tres genes está regulada por un puesto de control, lo que en inglés se llama un "checkpoint", pues no se inducen hasta que no comienza la segregación de los cromosomas. Si algo va mal en la replicación del DNA, por ejemplo tras la irradiación con luz ultravioleta, se inhibe la producción de dichas proteínas y se frena la división. También cesan de producirse cuando Sulfolobus no precisa dividirse, ya sea porque deja de crecer, se le inhibe con antibióticos que bloquean la división, o se le impide la división por medio de mutaciones.
Ni C ni Z, para nosotros división se escribe con S.
Resumiendo, los crenoarqueotas no poseen la exclusividad sobre su mecanismo de división celular. Lo que hacen es usar de forma distinta un mecanismo que los eucariotas conservan, pero dedican a otra función. Evidentemente eso tiene una serie de implicaciones filogenéticas. Si nos centramos en la filogenia vista desde el mundo de las arqueas, la primera divergencia se cree que ocurrió entre el linaje arquea/eucariota y el linaje bacteriano. Luego debió ocurrir la divergencia entre arqueas y eucariotas y finalmente la divergencia entre euryarqueotas y crenarqueotas. Estos resultados parecen indicar que el mecanismo de división basado en FtsZ es más antiguo, pero que quizás coexisitese con el mecanismo basado en CdvB y CdvC. Cuando sucedió la divergencia entre los euryarqueotas y los crenoarqueotas, los primeros continuaron con FtsZ y los segundos adoptaron el otro sistema. Los eucariotas sin embargo desarrollaron otro sistema de división celular y dejaron el sistema homólogo a Cdv para utilizarlo en la formación de vesículas, y a la heredera de FtsZ, la Tubulina, la colocaron en su citoesqueleto para llevar cosas, entre ellas los cromosomas, de un lado a otro de la célula.
Resulta muy interesante comprobar que en los organismos procariotas ambos mecanismos de división parecen excluirse entre si. Por un lado tendríamos el mecanismo de las bacterias, y euryarqueas que utilizan FtsZ. Por otro el que utilizan las creanarqueas, basado en las proteínas Cdv. Es decir, unas tienen motores de explosión y otras motores eléctricos. Pero parece que también existen los microorganismos, un grupo de crenarqueas y Thermoplasma acidophilum, una especie de euryarquea, que como los coches híbridos pueden funcionar con motor de explosión o eléctrico pues contienen tanto proteínas Cdv como FtsZ. Pero lo más chocante es que hay un tercer grupo de crenarqueas que no tienen ni proteínas Cdv ni FtsZ. ¿Usarán una turbina?

¿Eliges FtsZ o Cdv?
La distribución filogenética de los genes de división en arqueas indica que no es frecuente el caso en el que existen a la vez las proteínas Cdv y FtsZ. Los recuadros negros indican presencia de los genes, los números se refieren a los grupos filogenéticos a los que pertenece cada especie representada. Tomado del trabajo comentado.
REFERENCIA:
A-C. Lindås, E.A. Karlsson, M.T. Lindgren, T.J.G. Ettema, and R. Bernander. 2008. A unique cell division machinery in the Archaea. Proc Natl Acad Sci USA.
NOTA DE LOS AUTORES
Este artículo se publica simultáneamente en los dos foros administrados por cada uno de los dos autores, esta iniciativa es de esperar que anime al público de cada uno a visitar el otro para obtener una visión más amplia de las "curiosidades de esos pequeños bichitos ".
Este artículo ha sido traducido al inglés y publicado en el blog "Small Things Considered"